http://www.asyura2.com/20/senkyo270/msg/618.html
| Tweet | �@ |
http://www.asyura2.com/20/senkyo270/msg/607.html#c26�Ō������B
�����ɏ����Ă��鎖���������Ȃ�A����ȓ��{���ő厖�ɂȂ��Ă���Ƃ��ɁA����ȏd�v�ȃ}�j���A���ɊԈႢ�������Ă��A���̂܂ܓ˂��i��ł����̂����{�N�I���e�B���Ă��Ƃ݂������B����E���Ŕs��Ɍ������Ă܂������炩��B
�ȉ����p
http://iina-kobe.com/entry148/
���̃G���g����
�V�^�R���i�E�C���X�̑Ώ��@�Ƃ����ꂱ�ꏑ���Ă݂�
�V�^�R���i�E�C���X�̌����@�̊��x������ɂ��ď����Ă݂�
�_�C�������h�v�����Z�X���ɐ���������c���̓���̂��ꂱ���������Ă݂�
�̑����ł��B
�����́I
����́A�����Ȃ̏o�����a���̌��o�}�j���A���iRTPCR�̃}�j���A���j�ɂ��ď����܂��B
������Ɛ��I�ɂȂ�܂����A�������������̂�����ǂ��̂ł����A���������Ƃ���͂��������ƌ����Ă����Ȃ��ƃ_�����Ǝv���̂ŏ����Ă����܂��B
Contents
���J�Ȃ̃}�j���A���ɁA�v���I�Ȍ��ׂ�������܂����B2020.2.25�NjL
�����_���w�L�T�}�[�ƃI���SdT���g�p����Ă�����
���J�Ȃ̃}�j���A���̊�{�I�ȊԈႢ�������Ă����������̂ŁA�NjL���Ă����܂��B
3)�\1���߂����H pic.twitter.com/Hyhwdz23Y2
— TN (@tomoak1n) February 25, 2020
�Ȃ�ƁA����Ȃ��Ƃ������Ă����̂��킩��Ȃ��̂ł����A���[�o�[�X�g�����X�N���v�V�����iRT)�ɁA�A��G�̃��o�[�X�v���C�}�[�ł͂Ȃ��A�����_���v���C�}�[�ƃI���S��T�v���C�}�[���g�p����Ă���܂����B�B�B
�Ԃ����Ⴏ�A����ȏ����I�ȊԈႢ������Ƃ͎v���Ă��Ȃ��������߁A�������Ă���܂����B
�Ă�����A�펯�I�Ƀ��o�[�X�v���C�}�[��RT����Ă���Ǝv���Ă���܂����B�B�B�z��O�ł����B100���f�l���쐬�����}�j���A�����Ǝv���܂��B
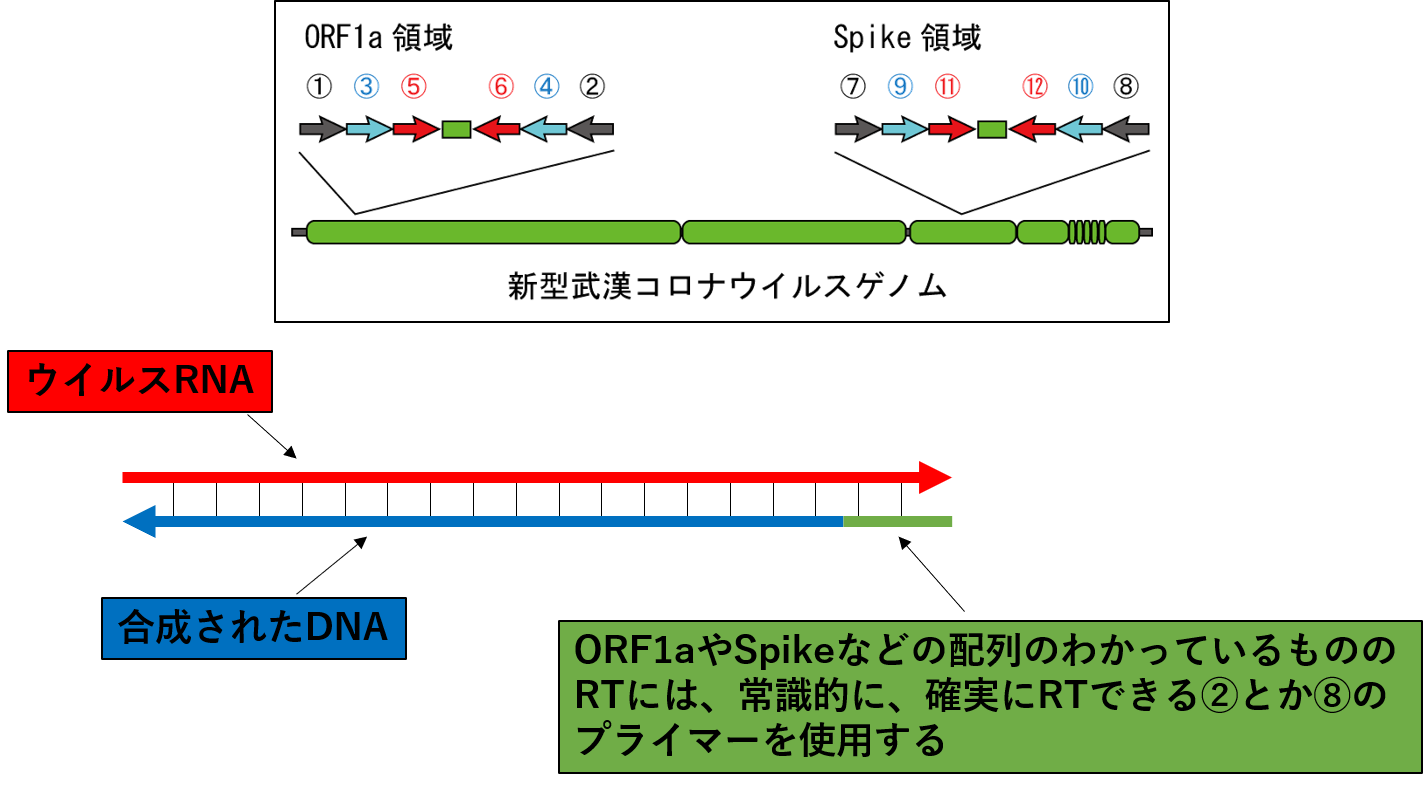
�펯�I�ɁAORF1a��Spike�Ȃǂ̔z��̂킩���Ă���RNA�̋t�]�ʁiRT�j�ɂ́A�m����RT�ł���A�Ƃ��G�̃v���C�}�[���g�p���܂��B
�������Ȃ���A���J�Ȃ�RT�̃v���g�R�[���ɂ́A�M�����Ȃ��ł����ARandom Hexamers ��Oligo(dT)12?18 Primer �Ƃ������̂��g�p����Ă���܂��B�B�B

�����_���w�L�T�}�[�Ƃ�
�����_���w�L�T�}�[�Ƃ́A���̖��̒ʂ�A�����_���Ȕz������v���C�}�[�̂��Ƃł��B
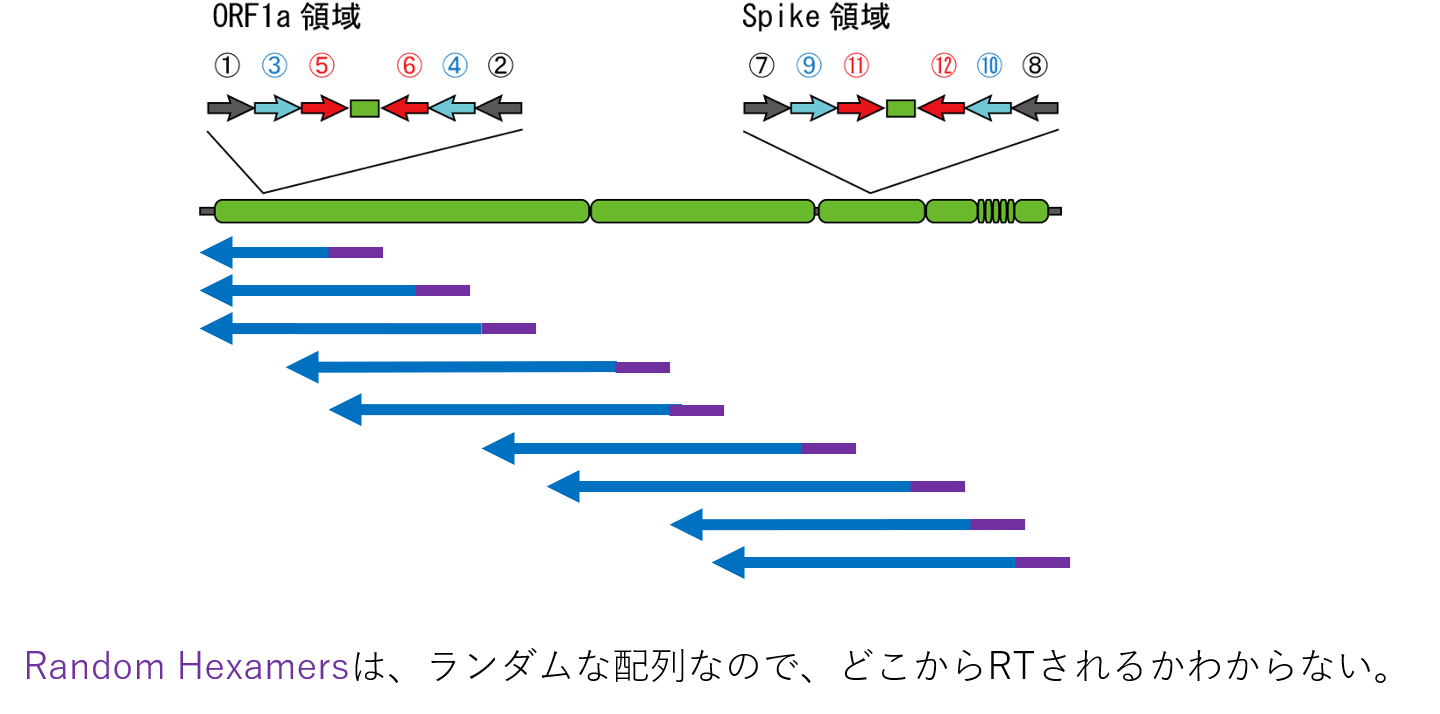
���̐}�̂悤�ɁARNA�Ƀ����_���ɂ������܂��B
���R�A�E�C���XRNA�ȊO�̊��җR����RNA�ɂ��������܂��B
�Ȃ̂ŁART�łł��Ă���cDNA�͂ȂɗR�����͂킩��܂���B
�܂��APCR����Ƃ��ɁA����������DNA�̈���܂�ł��Ȃ����̂���͓��RDNA�͑�������܂���B
�Ȃ̂ŁA�ʏ�͂���Ȃ��̂��g�p���܂���B
�܂��ART�̎��Ԃ�10���Ȃ̂ŁA�����_���v���C�}�[�����������Ƃ��납�璷����10kbp���������Ă��܂���B
�I���S��T�v���C�}�[�Ƃ�
�I���S��T�v���C�}�[�Ƃ́A�����T���A�Ȃ������̂ł��B
DNA��������mRNA�́A�����ۂ̕����Ƀ|��A�e�C���ƌĂ�邵���ۂ����Ă܂��B
�Ȃ̂ŁAmRNA���t�]�ʂ������Ƃ��ɂ����I���S��T�v���C�}�[���g�p����ƁA�|��A�e�C���ɂ������Ă���āA���ׂĂ�mRNA���t�]�ʂ��邱�Ƃ��\�ƂȂ�܂��B
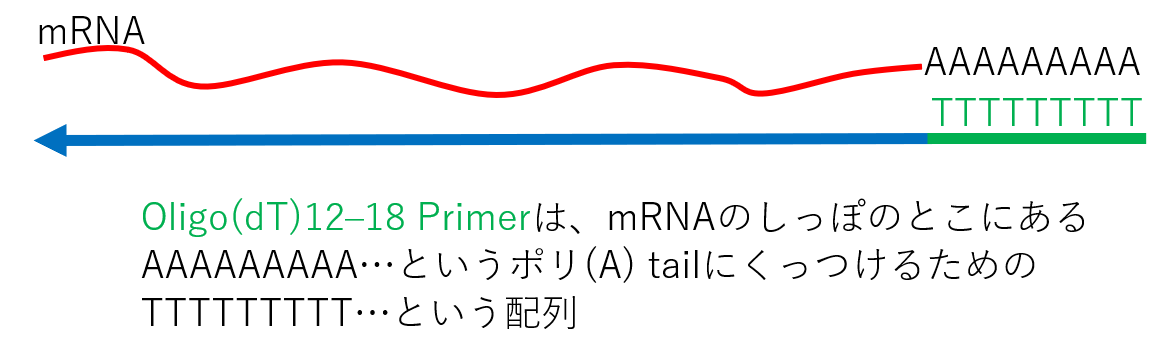
�Ȃ̂ŁA������E�C���X���ٓI��DNA����������̂ł͂���܂���B
�E�C���X��RNA���[�ɂ��|��A�e�C���͂���܂����A���������RT���s���ƁA�E�C���X�Q�m���̂����ۂ̕������獇�����n�܂�̂ŁA�Ō�܂ō����ł��Ȃ��ƁAPCR�ő����Ă��Ȃ��\��������܂��B
2019-nCoV�̉���z��
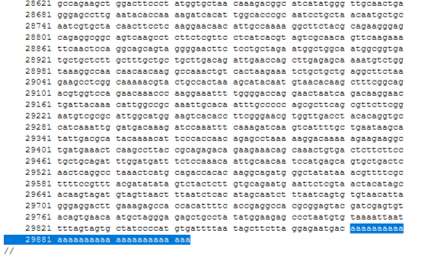
��
aaaaaa���ď����Ă���̂��|��A�e�C��
�܂��ART���s���|�������[�[��1���Ԃɖ�1,000bp�����̂ł����A2019-nCoV�̑S����29,903bp����̂ŁA���̃}�j���A���ɂ���10���ł́A10,000bp�܂ł��������ł����A�E�C���X��1/3�����Ƃ��Ă���܂���B
���R�Ȃ���AORF1a�܂ł͕����I�ɑ����Ă��܂���B
����āA���̃}�j���A���ɂ͒v���I�Ȍ��ׂ�����܂��B
���낤���đ����Ă��Ă�̂́A�����_���v���C�}�[�����܂��܋@�\���������ł��傤�B�B�B
���̃u���O�������������A����ɋC�t���Ă��Ȃ��������߁A�ȉ��̐����ł̓X�y�V�t�B�b�N�ȃv���C�}�[���g�p���������ŏ���������Ă��܂��B
�Ȃ̂ŁA���̂܂ܒu���Ă����܂��B�B�B
�{���̓X�y�V�t�B�b�N�ȇA�Ƃ��G�Ƃ��̃v���C�}�[��RT����̂������ł��B
���o���@
�E�C���X�����o����ɂ́A
1.���̂����B
2.���̂���RNA���Ƃ�B
3.RNA��RT���āARNA��DNA�̃n�C�u���b�h�����B
4.PCR�����đ��₷�B
�Ƃ����菇�݂܂��B
�iRTPCR�ɂ��Ă��V�^�R���i�E�C���X�̌����@�̊��x������ɂ��ď����Ă݂������ǂ݂��������j
���̎菇�ŁA
DNA�������Ă������z�����E�C���XRNA���聁�������Ă���
DNA�������Ă��Ȃ��������A�����E�C���XRNA�Ȃ����������Ă��Ȃ�
�ƂȂ�܂��B
���̎菇�ł́A�C��t���Ȃ��Ƃ����Ȃ��Ƃ��낪����������܂��B
���̂̎���
�܂��A���̂̃E�C���X���m���Ɏ�邱�����d�v�ł��B
����̐V�^�R���i�E�C���X�́A�x���������N�����܂��̂ŁA�x�̒���C���̉��̕��ő�����Ǝv���܂��B
�Ȃ̂ŁAႁi����j��A�C���̉��̔S��������Č��̂Ƃ���̂��d�v�ł��B
�A�ɂ͂���܂肢�Ȃ����ۂ��̂ŁA�A���炾�Ƃ����Ԃ�E�C���X�������Ă��炶��Ȃ���RNA�͎��Ȃ��Ǝv���܂��B
����A�ŏ��͉A�����ォ��z�����ĂȂ�̂́A�A�������Ă邩�炾�Ǝv���܂��B
�E�C���X������̂��E�C���X�������Ă��Ȃ����̂����̂Ƃ��ĂƂ����Ⴄ�ƁA�ǂ�Ȃ��Ƃ��Ă��E�C���X�����Ȃ��̂Ō��o�ł��܂����B
�Ȃ̂ŁA���̂̍̎���@�͍ŏd�v�ł��B
RNA�̎�舵�����@
RNA���Ă̂́A���ɉ��₷���ł��B
�Ȃ̂ŁA����Ȑl�����ƁARNA�����Ă��܂��\��������܂��B
���x���������������A���Ԃ�������������肷��Ƃǂ�ǂ�RNA�����Ă������Ⴄ�̂ŁA�f�������邱�Ƃ��厖�ł��B
��������͋Z���̘r����ł��B
���Ȃ݂ɁA����̃}�j���A���ł͕K�v�̂Ȃ��Ǝv����菇�������������̂ŁA���̎菇�ł��ƌ��\����Ǝv���܂��B
PCR�̐��x
������RNA�����Ă��Ă��A�v���C�}�[�̐v���_���_������DNA�������ɂ����ł��B
�v���C�}�[�̐v�͔��ɑ厖�ł��B
�v���C�}�[�̐v�ɂ͏n���̋Z���K�v�ł��B
�v���C�}�[���ǂ���Ή��肭�������Ă��@�B�����₵�Ă���܂��B
���x���R�O���`�T�O���ƌ����Ă��闝�R�́ARNA�����܂����Ă��Ȃ��̂ƁA�v���C�}�[�v�����肭���Ȃ̂��������Ǝv���܂��B
RTPCR����ꏊ
�}�j���A���ɂ́A
�V�^�R���i�E�C���X�i2019-nCoV�j����`�q�̈�Q�����Aopen reading flame 1a (ORF1a) ����� spike (S)����ٓI�Ɍ��o���� 2-step RT-PCR �@�A���邢�� TaqMan �v���[�u��p�������A���^�C�� one-step RT-PCR �@�ɂ���`�q�����ɂ�� 2019-nCoV �肷��B
�Ə�����Ă���܂��B
�ǂ������Ӗ����Ƃ����ƁA�V�^�R���i�E�C���X���L�̓�̏ꏊ��RTPCR�ő��₵�āA���ꂪ�V�^�R���i�E�C���X���ǂ����ׂ܂��傤�B���Ă��Ƃł��B
2-step RT-PCR �@�ɂ��Ă͂��Ƃʼn�����܂��B
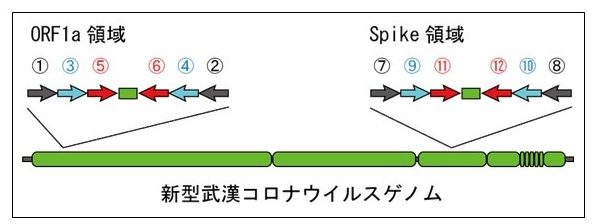
��`�q�̈�̓�̂����̈�ڂ́@open reading flame 1a (ORF1a)���ĂƂ��ł��B
ORF1a�́A�I�[�v�����[�f�B���O�t���[���@�����G�[�@�Ɠǂ݂܂��āA�E�C���X�̈�`�q���R�[�h���Ă��镔���̈�ԖڂƂ������ƂɂȂ�܂��B
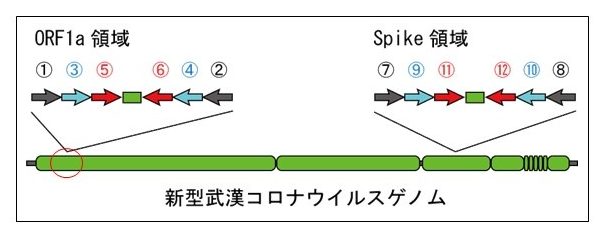
���̏�̐}�ł����A�Ԋۂň͂����Ƃ���ւ�ł��B
���̐}�ł����A�R�̂Ƃ���PLpro���ď����Ă�Ƃ��낾�Ǝv���܂��B
���Ȃ݂ɁA����A���J�Ȃ̂������}�j���A���Ȃ�ł����A�V�^�����R���i�E�C���X�Q�m�����ď����Ă�̂ŁA�����R���i�E�C���X���Ă����ȁ[�I���Č����Ă�l�́A�܂����J�Ȃɕ��匾���܂��傤�B
�����Ă�����Aspike (S)�́A�X�p�C�N�̂���ς������R�[�h���Ă���Ƃ��ł��B�X�p�C�N�̓R���i�E�C���X�̃M�U�M�U�̕����ł��ˁB
����́A���̐}�ł����ۂ̂Ƃ��ł��B
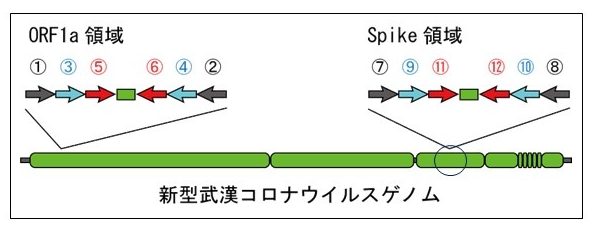

���̏�̐}�ł���S���ď�����Ă���Ƃ��ł��ˁB
�����̗̈悪�A�u�V�^�����R���i�E�C���X�ɓ��L�Ȕz���v�炵���̂ŁA�����̔z���RTPCR���������Ă���A�V�^�R���i�E�C���X��RNA�������I���z������I���������Ă����I���Ă��ƂɂȂ�܂��B
RT�p��primer�̐v
���o�[�X�g�����X�N���v�V����
���āA���₵���ł����A�V�^�R���i�E�C���X�̌����@�̊��x������ɂ��ď����Ă݂��ł��Љ�܂���RTPCR���Ă������̂��g���čs���܂��B
����RTPCR�ň�ԏd�v�ɂȂ��Ă���̂́A�v���C�}�[�̐v�ł��B
�v���C�}�[���Ă̂́A�V�^�R���i�E�C���X��RNA�̔z��ɂ҂����ȁi����I�ȁj�z�������20����炢��DNA�ŁA�l�H�I�ɍ������č��܂��B
���������ꖜ�A1,000�~���炢�ł��B
���̃v���C�}�[���A�V�^�R���i�E�C���X�ɓ��L�Ȕz��Ƀs���|�C���g�ł�������ƁA������DNA�𑝂₷�N�_�ƂȂ�܂��B
�܂��ŏ��ɁA�E�C���X��RNA�ɂ�������v���C�}�[�́A���̐}�ł����A�ƇG�ɂȂ�܂��B
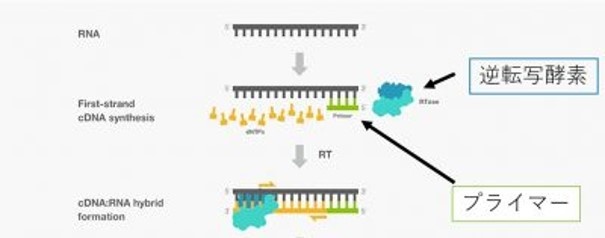
���́A�A�ƇG�̃v���C�}�[�i���������j�ŁA���ꂼ��RT������Ă��Ƃł��ˁB
���̃v���C�}�[�̔z���
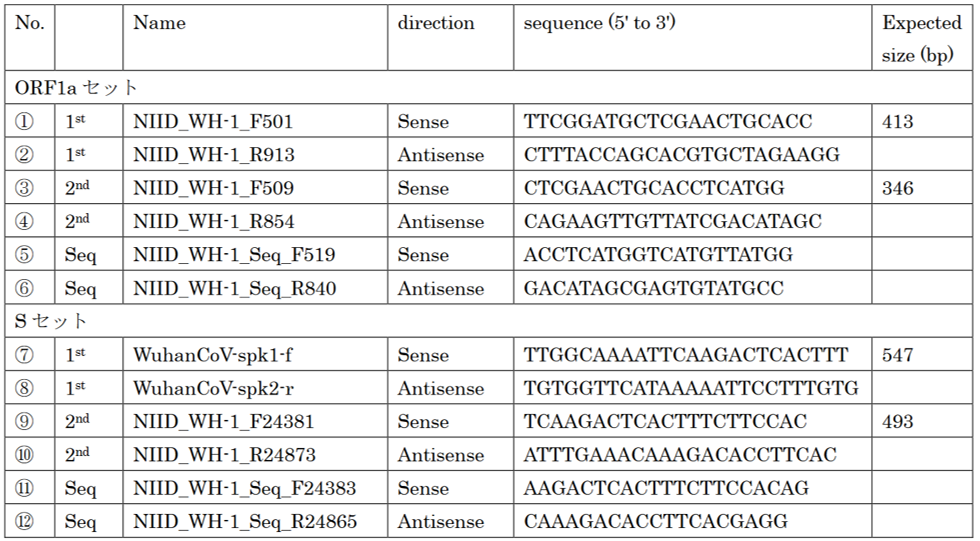
�̇A�ƇG�ł��ˁB
ORF1�𑝂₷���߂ɍs��RT�Ɏg���v���C�}�[���A�́@CTTTACCAGCACGTGCTAGAAGG�@�ł��B
�X�p�C�N�𑝂₷���߂ɍs��RT�Ɏg���v���C�}�[���G�́@TGTGGTTCATAAAAATTCCTTTGTG�@�ł��B
����Ń��o�[�X�g�����X�N���v�V�����iRT)������ƁA���̐}�̂悤�ȁ@�E�C���XRNA�{�v���C�}�[���N�_�Ƃ����E�C���X��RNA�ɂ҂����肭����DNA�́@RNA/DNA�̓�{�����ł��܂��B

�����̂���
���J�Ȃ̏o���Ă���RT�̂����́A
23��C�@10min
50��C�@10 min
80��C�@10 min
���ď����Ă���܂����A������ƈӖ���������܂���B
23�x��10���ɉ��̈Ӗ�������̂��킩��܂���B
�ӂ��́A65����5���@���@4����1���Ɣ��������A�v���C�}�[��RNA�ɂ����������܂��B
���̌�A50����10�������āA�t�]�ʍy�f��DNA�����������܂��B
DNA������A80���ŋt�]�ʍy�f�������i�j�����܂��B
�������邱�Ƃ�RNA/DNA�̓�{�����ł��܂��B
�ŏ��Ɉ��65���ɉ��x���グ�Ȃ��ƁA�v���C�}�[���������ɂ����Ǝv���܂��B
�Ȃ��Ȃ�A�v���C�}�[��DNA��RNA�ɂ��������x���Ă����̂������āiTM�l���Ă����܂��j���̉��x�ɂ��Ă����Ȃ��Ƃ������Ȃ�����ł��B
TM�l�́A�iA+T�̐��jx2�{�iC�{G�̐��j��4�|�iA+T+C+G�̐��jx2-35�ŋ��߂邱�Ƃ��ł��܂��B
�i����́A���̏ꍇ�ł��B�l�ɂ���ĈႤ�Ǝv���܂��B�j
����̏ꍇ�A�A�̃v���C�}�[��TM�l�́iA+T�̐��jx2�{�iC�{G�̐��j��4�|�iA+T+C+G�̐��jx2+35��11�~2+12�~4-23�~2+35��59��
�G�̃v���C�}�[��TM�l��17�~2+8�~4-25�~2+35��51��
�ł��B
�Ȃ̂ŁA��x65���ɂ����Ă����āA4���ɗ�₷�ߒ��ŁA59����51���ɗ₦���Ƃ��Ƀv���C�}�[�����������Ă��ƂɂȂ�܂��B
�����23����10����闝�R���킩��܂���B
�����āA�t�]�ʍy�f�̊�����45���`50������ԍ����̂ŁA50����DNA������������̂ł����A59���̃v���C�}�[�͂��̕��@�ł�RNA�ɂ������Ȃ��Ǝv���܂��B
RNA/DNA�̓�{���̉��
RT���I�������A�ł���RNA/DNA�̓�{����������܂��B
RT���Ă����̂́A���X�������E�C���XRNA�̐�����RNA/DNA�̓�{���̐��͂ł��܂���B
10��RNA����������A�ō��ł�10��RNA/DNA�̓�{�������ł��܂���B
�Ȃ̂ŁA���̗n�t���ł��邾����������g����PCR�����邱�ƂŁA�����悭DNA�����邱�Ƃ��ł��܂��B
�������Ȃ���A���J�Ȃ̃}�j���A���ł́A�Ȃ������������ł���RNA/DNA�̓�{���𔖂߂Ă��܂��B
��

25�ʂ��̔����n��RT���s������A�Ȃ������iDDW)��35�ʂ������āA60�ʂ��ɂ��āA���̂�����5�ʂ��������A����PCR�Ɏg���Ă��܂��B
�Ⴆ�A25�ʂ���RNA/DNA�̓�{����25�{�ł��Ă����i�E�C���X��25�������j�Ƃ���ƁA2�{�ɂ܂Ō��炵������Ă邱�ƂɂȂ�܂��B
������������25�ʂ��̂�����5�ʂ���PCR�ɂ��̂܂g����5�{�����Ă���̂ɁA�Ȃ������߂Č��o���x�����炵�Ă��܂��B
�Ȃ��Ȃ̂��͂킩��܂���B
PCR
�C����蒼����PCR�̉���ɍs���܂��B
�|�W�R���ƃl�K�R��
PCR���Ă̂́A�{���ɃE�C���X�������猟�o�ł���B�Ƃ����|�W�e�B�u�R���g���[���i�|�W�R���j�ƁA��ɃE�C���X�͌��o����Ȃ���I���Ă����l�K�e�B�u�R���g���[���i�l�K�R���j�̓���K�v�ł��B
�Ȃ��Ȃ�A��ɏo�����o�Ȃ�������A�o�Ȃ�����o�Ă����肷��Ƃ��������PCR���̂����s������A�Ӗ����Ȃ�����ł��B
���̃|�W�e�B�u�R���g���[���ɂ́A�V�^�����R���i�E�C���X�����邱�Ƃ��m�肵�Ă������̂��瓯���悤��RT����RNA/DNA�̓�{�����g�p���܂��B
�l�K�e�B�u�R���g���[���ɂ́A�V�^�����R���i�E�C���X���Ȃ����Ƃ��m�肵�Ă������̂��瓯���悤��RT����RNA/DNA�̓�{�����g�p���܂��B
�Ȃ̂ŁA�����A���Ȃ��Ƃ��A�|�W�R���A�l�K�R���A���ׂ�����@�̎O��RT���Ă����K�v������܂��B
�������Ȃ����A���J�Ȃ̃l�K�R���́A�����̐����g�p���Ă���܂��B

���A���ΏƁi�l�K�R���j�Ƃ���DDW�i�������j 5��l��p����
��Ȃ́A���Ȃ�Ďg������100���łȂ�����A�l�K�R���ɂ͂Ȃ�Ȃ��ł��B
�������ł�����l�K�R�����Č������珬�ꎞ�Ԑ������炤���x���̂���Ă͂����Ȃ������ł��B
PCR�̃v���C�}�[�v
��قǂ��������悤�ɁA�v���C�}�[��DNA��RNA�ɂ��������x�͂��̃v���C�}�[��TM�l�i�A�j�[�����O���x�j�Ɉˑ����܂��B
�����pcr�́A�@�̃v���C�}�[�ƁART�Ŏg�����A�̃v���C�}�[���g�p���܂��B
�@�̃v���C�}�[�̔z��́A��ɂ������\����A�@TTCGGATGCTCGAACTGCACC�@�ŁA����TM�l��9�~2+12�~4�|21��2+35��59��
�A��TM�l��11�~2+12�~4-23�~2+35��59��
�Ȃ̂ŁA���̓�̃v���C�}�[�́A59����DNA�ɂ��������Ă��Ƃ��킩��܂��B
�Ȃ̂ŁAPCR�̂����Ƃ��ẮA95���œ�{�����͂����Ă���āA59���Ńv���C�}�[���A�j�[�����O�i��������j���āA72���ŐL�������iDNA����点��j���Ă����T�C�N�����A��������35�T�C�N�����炢���̂������Ǝv���܂��B
���J�Ȃ̏o����PCR�̂����́A94��C�@30sec�@�œ�{�����͂����A56��C�@30sec�@�ł������A68��C�@1min�ŐL��������40�T�C�N�����ď����Ă���܂��B
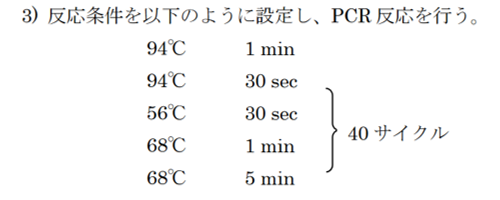
�܂��A����ł��ł���ł��傤�B�B�B
�����Ȃ݂ɁA�L��������68���ł��̂́A�A�j�[�����O���x��68�����炢�ɐݒ肵���v���C�}�[���g�p���āA�A�j�[�����O�ƐL����������C�ɂ���Ă��܂����I���Ă��������e�N�j�b�N�Ȃ̂ł����A68�����ƐL�������̊�����������̂ŁA���ʂ�72���ł��܂��B
���́A�X�p�C�N��RNA�𑝂₷�F�ƇG�̃v���C�}�[���g����PCR�ł��B
�F�̃v���C�}�[�̔z��́ATTGGCAAAATTCAAGACTCACTTT�ŁATM�l�́A16�~2+8��4-24��2+35��51��
�G�̃v���C�}�[��TM�l�͂������v�Z�����ʂ�A51���ł��B
����PCR�̏ꍇ�A
�A�j�[�����O�̉��x��51�x�܂ʼn����Ă����Ȃ��ƁA�v���C�}�[��DNA�ɂ��������Ƃ��ł��܂���I
�Ȃ̂ŁA94��C�@30sec�@�œ�{�����͂����A56��C�@30sec�@�ł������A68��C�@1min�ŐL��������40�T�C�N�����Ă������@�ł́A�v���C�}�[��DNA�ɂ������Ȃ��̂ł��B
�Ȃ̂ŁA�����܂���B
������A���s����ł��傤�B
���ł��A���̂�������PCR������܂���B
��������S�Ƀv���C�}�[�̐v�~�X�ł��B
2-stepRTPCR�@�inestedPCR)
���āA���̐}�̐Ԋۂ̒ʂ�A��L�̇@�ƇA�̃v���C�}�[�̑g�ݍ��킹�ŁA���܂������ƁA413bp�i413����̒����j��DNA����������Ă��܂��B
�F�ƇG�̑g�ݍ��킹���ƁA547bp��DNA����������Ă��܂��B
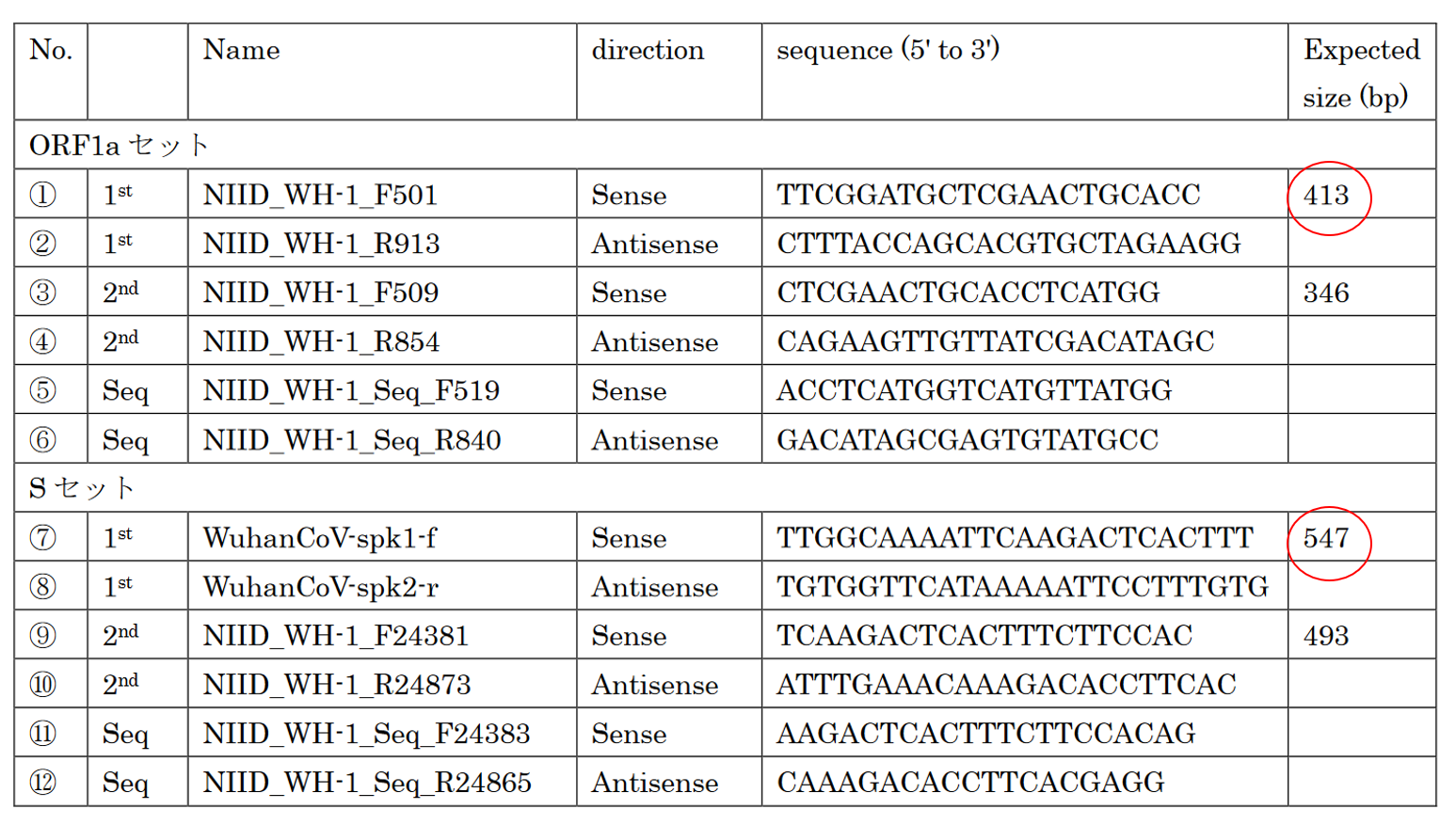
���̑�������Ă���DNA�́A���X�̓E�C���X�R���̂��̂Ȃ͂��ł��B
�Ȃ̂ŁA
�@�ƇA�̃v���C�}�[�̑g�ݍ��킹�łł���413bpDNA�͇@�ƇA�ŋ��܂ꂽ�̈�̔z�܂܂�Ă���͂��ł��B
�Ȃ̂ŁA�B�ƇC�̃v���C�}�[��PCR������A346bp��DNA�����������͂��ł��B
�����A�����Ă����̂��S���Ⴄ���̂��Ƃ�����A�B�ƇC�̃v���C�}�[�͔z�F���ł��Ȃ��̂ŁADNA�͑����Ȃ��͂��ł��B
���́A�Q�i�K�ł�PCR�`�F�b�N��2-stepRTPCR�@�������́Anested�@PCR�Ƃ����܂��B
����́A���ِ������m���߂邽�߂ɍs���܂��B
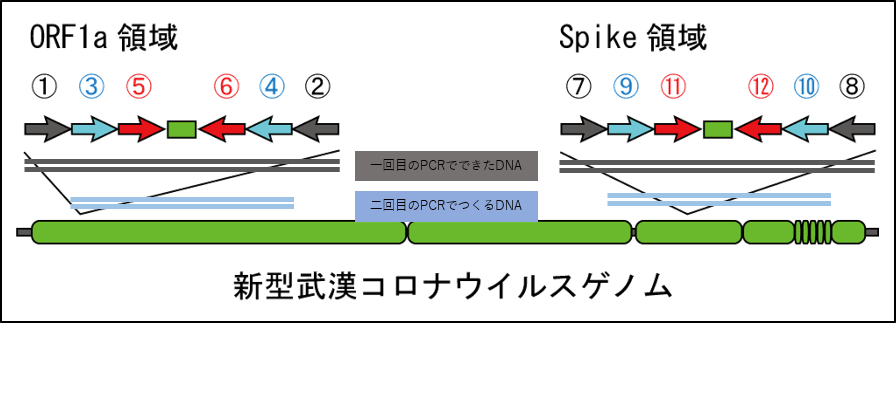
�F�ƇG�̃v���C�}�[�̑g�ݍ��킹�łł���547bp��DNA�̓����ɐv���ꂽ�H�ƇI�̃v���C�}�[�ł́A493bp��DNA����������܂��B
�������Ȃ���A
�B�̃v���C�}�[�̔z��́@CTCGAACTGCACCTCATGG�@�Ȃ̂ŁATM�l��8��2+11��4-19��2+35��57��
�C�̃v���C�}�[�̔z��́@CAGAAGTTGTTATCGACATAGC�@�Ȃ̂ŁATM�l��13��2+9��4-22��2+35��53��
�Ȃ̂ŁA�A�j�[�����O���x��56���̂��̕��@����A�C�̃v���C�}�[���A�j�[�����O�ł��Ȃ�����A�����܂���B
�H�̃v���C�}�[�̔z��́@TCAAGACTCACTTTCTTCCAC�@�Ȃ̂ŁATM�l��12��2+9��4-21��2+35��53��
�I�̃v���C�}�[�̔z��́@ATTTGAAACAAAGACACCTTCAC�@�Ȃ̂ŁATM�l��15��2+8��4-23��2+35��51��
�Ȃ̂ŁA�A�j�[�����O���x��56���̂��̕��@����A�H�̃v���C�}�[���I�̃v���C�}�[���A�j�[�����O�ł��Ȃ�����A�����܂���B
��������A�v���C�}�[�̐v�~�X�ł��B
���ƁA�l�X�e�B�b�hPCR�́A25�T�C�N�����Ώ\���Ȃ̂ɁA�Ȃ���40�T�C�N�����Ă���͖̂��ʂł��B
�Ȃ�Ƃ����肬��łłĂ���PCR�Y�����A�X���A�i�������������Ă���j���Ă��Ă��̎��_�Ń_���ł��ˁB��
���Ȃ݂ɁA����20�N�ȏ�O�ɂ������ł����ꂭ�炢��������o�܂��B�����͈�掆�Ɉ����������ł����A����ł����̃��x���ł��B
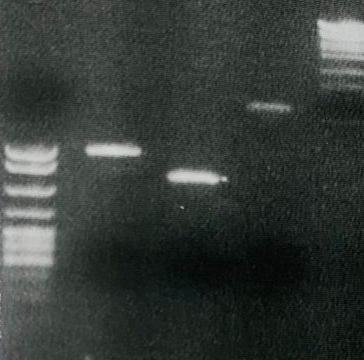
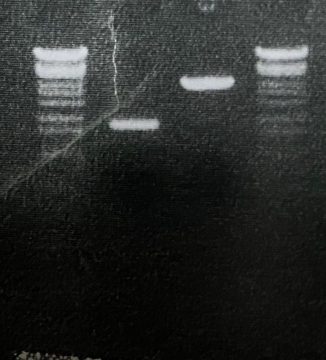
�܂��A����Ŋ��x��30���`50���ƌ����Ă��A����ᓖ����O����˂��B�B�B�Ƃ����v���܂���ˁB�B�B
���̐��x���Ⴂ�̂́A�܂��A�܂ŃE�C���X���ĂȂ��̂ɍA�̔S������ăT���v���ɂ��Ă�����ARNA���o�����肷���ĉ�ꂿ������肵�Ă���Ǝv���܂��B30?50%��PCR���x���āA�w�������x�����ƁB�B�B
— ������ (@iina_kobe) February 10, 2020
���A���^�C�� one-step RT-PCR �@
���J�Ȃ́ATaqMan�v���[�u��p�������A���^�C�� one-step RT-PCR �@���Ă������̂��Љ�Ă��܂��B
����͉����Ƃ����܂��ƁA���ʂ�PCR�ɁATaqMan�v���[�u�Ƃ������̂������čs��PCR�ł��B
TaqMan�v���[�u�́A�v���C�}�[�Ɠ����悤�ɁA�W�I��DNA�i����̏ꍇ�́A�E�C���XRNA��RT���č��ꂽDNA�j�ɂ��������̂ł��B
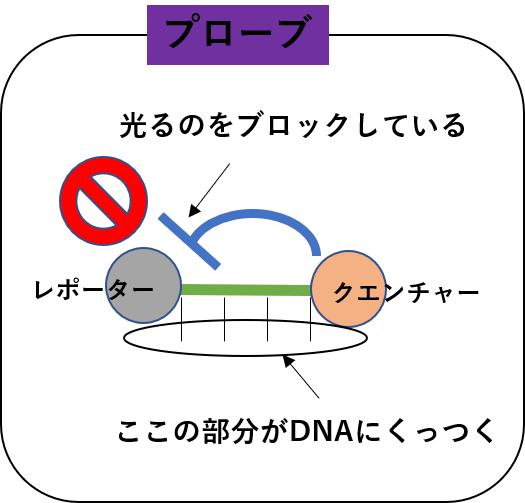
�v���[�u�́A�v���C�}�[�̗��[�ɁA���|�[�^�[�Ƃ����u�����镔���ƁA�N�G���`���[�Ƃ������ꂪ����Ȃ��悤�ɂ��镔���Ƃ����������悤�Ȍ`�����Ă��܂��B
���ꂪ�ART�łł���DNA�ɂ������Ă��܂��B
PCR���s���ƁA�v���C�}�[���N�_��DNA����������Ă����܂��B
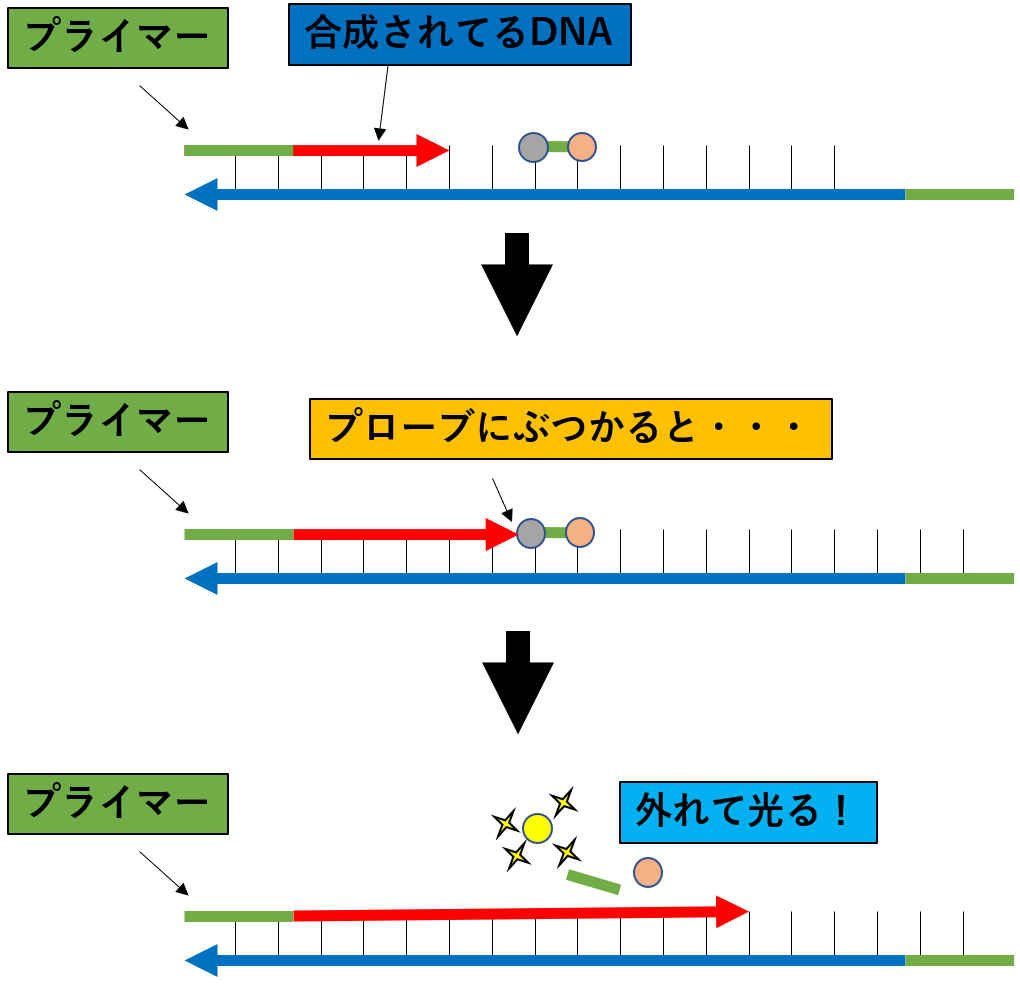
�������v���[�u�����������Ƃ���܂Ői�ނƁA�v���[�u�͉����̂����ĕ������Ă��܂��܂��B
���̎��A���|�[�^�[�͌���Ȃ��悤�Ƀu���b�N���Ă����N�G���`���[�Ƃ̋������A�����悤�ɂȂ�u�����܂��B
����āA�u�������o�������A����DNA�����ꂽ���E�C���X�R����DNA���������Ƃ������ƂɂȂ�܂��B
���̗��_�́APCR���Ɍu�������o���鑕�u�Ŋm�F�ł��邽�߁ADNA��������Α�����قnju���������āA�ǂꂭ�炢�������������A���^�C���ł킩��Ƃ��������b�g������܂��B
�Ȃ̂ŁAReal time(���A���^�C��)PCR�ƌ����Ă��܂��B
���A���^�C���ƁA���o�[�X�g�����X�N���v�V���i���͂ǂ�����RT�Ɨ������߁A��ʂ̐l�͍������Ă��܂��܂��ˁB
���āA���̎�@���g�������J�Ȃ̃}�j���A���ɂ́A
95��C�@15sec.
60��C�@60sec.(Data Collection)
�Ƃ������̂�45�T�C�N���B���ď����Ă���܂��B
������A�Ȃ�����Ȃ��ƂɂȂ����̂��Ӗ��s���ł��B
���Ȃ݂ɂ��̎��Ɏg���Ă���v���C�}�[��1�Z�b�g�ڂ�
CACATTGGCACCCGCAATC�@��TM�l�@11��4+8��2-19��2+35��57��
GAGGAACGAGAAGAGGCTTG�@��TM�l�@11��4+9��2-20��2+35��57��
�Ȃ̂ŁA60��C����A���x���������ăv���C�}�[��DNA�ɂ������܂���B
60������ADNA�̐L���������������Ǝv���܂��B
�v���C�}�[�Q�Z�b�g�ڂ�
AAATTTTGGGGACCAGGAAC�@��TM�l�@9��4+11��2-20��2+35��53���@
TGGCAGCTGTGTAGGTCAAC�@��TM�l�@11��4+9��2-20��2+35��57��
�Ȃ̂ŁA������܂�60��C����A���x���������ăv���C�}�[��DNA�ɂ������܂���B
�v���C�}�[�̐v�~�X�ł��B
���U�O���ŃA�j�[�����O�ƐL���������s���̂́A�v���[�u���͂���Ȃ��悤�ɂ��邽�߂Ƃ̌���������܂����A���������������鉖������Ȃ��i158bp�j�ׁA�͂����O�ɕ����ł���Ǝv���܂��B
���Ȃ݂ɁA�����̃}�j���A���́A
���M�҈ꗗ
���ˌ���@�b��w���m(�k�C����w)�@�������@���R�B���@�|�c���@(���������nj������E�C���X��O��)
�e�R�w�@����w��w�� ��w���m�i�����������������C���t���G���U�E�C���X�����Z���^�[�j
���P���@�R����w ��w���@�i�R�������ی��Z���^�[�j
�l�{���l������ȑ�w, ��w���@�i���Q�����q�����������j
�炪�����ꂽ���̂炵���ł��B
�S���b��w���������͈�w���ł����ˁB�B�B
�����_�����B�B�B
���Ȃ炱������I
�����̍��ڂ͎��̃G���g���ɉ��߂ď������Ƃɂ��܂����̂ŁA�폜���Ă����܂��B
2020.2.28�@11�F16
 �X�p�����[���̒����猩���o�����߂Ƀ��[���̃^�C�g���ɂ͕K���u���C������ցv�ƋL�q���Ă��������B
�X�p�����[���̒����猩���o�����߂Ƀ��[���̃^�C�g���ɂ͕K���u���C������ցv�ƋL�q���Ă��������B